针对人脑肿瘤的深度空间蛋白组学检测
针对人脑肿瘤的深度空间蛋白组学检测
细胞组成和组织的空间组织对其特性和功能起着至关重要的作用。了解这些特征对于研究疾病结局至关重要。最近在空间分辨测序技术方面的进展使得我们能够对组织中的基因表达模式进行表征。然而,要充分理解组织的异质性,还必须考虑基因所编码的蛋白质。质谱成像(MSI)技术可以在组织中绘制蛋白质分布,但在生成全面的蛋白质组数据方面存在局限性。激光捕获显微取样(LCM)作为一个有希望的解决方案,可以从细胞单元到更大的组织区域对组织中的特定区域进行提取。
最近,来自牛津大学的Simon Davis等人使用激光捕获显微取样技术分析了人类脑肿瘤的蛋白质组,分辨率从整个组织切片到40微米。主要目标是开发一种深度蛋白质组学和空间感知统计学的工作流程,以发现与组织特征相关的内容,而不是专注于脑肿瘤的具体生物学。通过应用空间感知统计方法,他们在肿瘤切片中确定了具有差异空间和聚类表达的蛋白质和通路,在没有组织结构或病理学的先验知识的情况下。此外,他们在肿瘤中发现了具有空间定义的蛋白质表型,揭示了具有空间分辨的细胞外基质生物学和肿瘤边缘的聚焦免疫应答。此外,他们以与血管细小结构相关的蛋白质丰度为基础,提供了有关癌组织内营养和氧气依赖的空间蛋白质组的见解。这项研究展示了深度地形蛋白质组学与空间感知分析相结合的潜力,以识别与疾病机制相关的感兴趣区域。
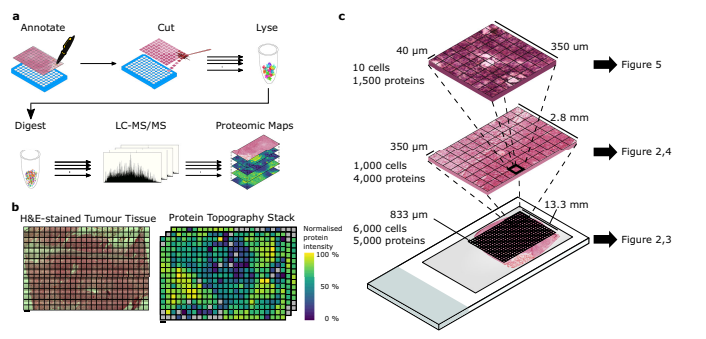
他们的工作方法
从组织供体获得了经过适当同意的尸体脑组织,并存储在牛津脑库。大脑切片并从第一和第二冠状切片中分离出肿瘤组织。从肿瘤的不同部位取了冷冻切片以确定保存最佳的切片。选择第二冠状切片的右上象限进行进一步实验。将组织块放在载玻片上,并用H&E染色。准备好进行激光捕获显微取样的组织块。
使用配备了激光压力弹射设备的激光捕获显微镜进行组织分析,具体是PALM Microbeam和Leica LMD7系统。PALM Microbeam用于10倍物镜分析,而LMD7用于20倍物镜分析。微光束的切割和捕获参数设置为切割能量:43,焦点:55,捕获能量:20,焦点:-15。从两个系统收集的样品存放在-80°C,待进一步使用。对于LMD7系统,组织区域被显微取样并转移到96孔PCR板中,经以乙腈处理,然后在-20°C储存待进一步处理。
样品解冻后,简短离心并用含苯佐卡因的RIPA缓冲液冲洗以收集任何残留组织。然后将蛋白质还原和烷基化后与顺磁性SP3磁珠混合。样品经过乙腈处理并震荡以使磁珠定位于磁铁上。洗涤后,重新悬浮磁珠,用胰蛋白酶消化过夜,然后超声处理。重复进行乙腈处理和磁珠定位,然后将肽转移到样品瓶或板上。对于40微米分辨率的数据,使用n-十二烷基-β-D-麦芽糖苷和胰蛋白酶/ LysC混合消化缓冲液进行一步消化。利用C18尖端进行LC-MS / MS分析加载产生的肽。
使用不同的LC-MS / MS系统分析具有833微米,350微米和40微米分辨率的样品肽。对于833微米分辨率,使用Dionex Ultimate 3000与timsTOF Pro相结合,配备75 μm x 150 mm C18柱和流量为400 nL / min。应用17分钟的线性梯度,从2%的缓冲液B到30%的缓冲液B,质谱图谱在100-1700 m/z范围内记录。对于350微米分辨率,使用Dionex Ultimate 3000与Orbitrap Fusion Lumos相结合,配备75 µm x 500 mm C18柱和流量为250 nL / min。应用60分钟的线性梯度,从2%的缓冲液B到35%的缓冲液B,MS1扫描在400-1500 m/z范围内记录。至于40微米分辨率,使用Evosep One LC系统与timsTOF SCP质谱仪相结合,配备75微米x 150 mm C18柱,每个周期的流速为1.07秒。将diaPASEF帧分成3个离子迁移窗口,涵盖400-1000 m/z的质量范围。
他们发现了什么
研究人员描述了在非典型畸胎-梭形肿瘤(AT / RT)上进行空间分辨蛋白质组学的工作流程。他们获得了肿瘤块的10微米厚切片,并将其分为384个正方形“体素”进行分析。使用激光捕获显微取样(LCM)将每个体素分离并经过其LCMSP3方案处理。他们成功鉴定出5321种蛋白质,每个样本中鉴定出的蛋白质数量各不相同,包括空的体素。然后将识别出的蛋白质映射回其原始在组织网格内的位置。展示了四种示例蛋白质在肿瘤切片中具有不同的表达模式。对4306种定量蛋白质进行了空间自相关分析,结果显示3212种蛋白质呈现出相关的空间表达。研究人员确保没有采样偏差,并且蛋白质丰度差异主要是组成性的。为了提高空间分辨率,他们聚焦于肿瘤内的特定区域,并在96个较小的体素中量化了3994个蛋白质。这个区域中四种示例蛋白质的表达模式与更大视野的数据一致。他们还使用免疫组织化学染色对空间分辨蛋白质表达数据进行了进一步验证,选取了三种特定蛋白质。染色模式与蛋白质密度分布在蛋白质组学地图中基本一致。
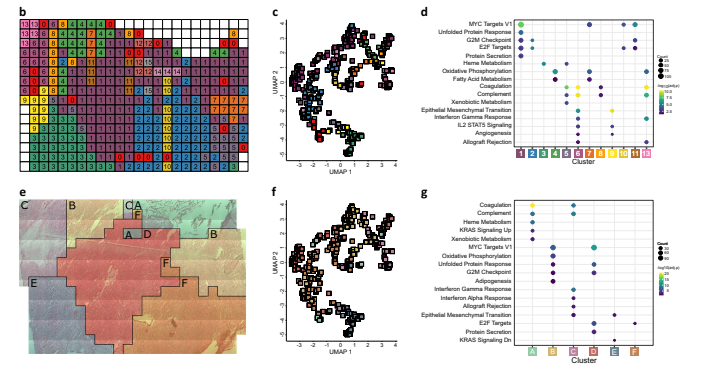
AT / RT中固体肿瘤和脑部/肿瘤界面之间的边缘采用聚类方法进行分析,基于蛋白质定量数据生成不同的空间定义区域。这种方法鉴定的聚类与UMAP可视化中观察到的空间聚类一致。确定了每个聚类簇的标记蛋白质以研究其功能差异。代表固体肿瘤的聚类1和2显示增殖标志物的富集,而相邻的聚类6和13位于左上区域,富集免疫相关标志物。位于肿瘤边缘的聚类9显示富集与血管生成和上皮间质转化相关的标志物,表明活跃的肿瘤浸润。免疫细胞标记蛋白质的分析显示了中性粒细胞标记物和促肿瘤M2巨噬细胞标记物在聚类13和6中的定位。使用综合蛋白质组学和空间相似性方法进行空间聚类得到了六个聚类,涵盖了固体肿瘤、脑部/肿瘤界面、免疫浸润和出血。这些聚类在与其相关功能相关的特定标志物方面显示出富集。这项研究中发现的聚类结果在不同的空间分辨率下分析时保持一致。总体而言,这些研究中发现的聚类有助于确定AT / RT中肿瘤边缘的功能层次,突显了固体肿瘤、界面和周围区域之间的蛋白质丰度和功能过程的差异。
研究人员专注于肿瘤组织中的一个特定区域,该区域包含一个由单个体素表示的四个血管。他们使用更高的空间分辨率350微米分析蛋白质丰度模式及其与营养和氧梯度的关系。该区域进一步划分为9 x 9的网格,得到40微米的空间分辨率。每个40微米的体素包含5到10个可见细胞核,共计1550种蛋白质使用数据无关分析进行定量。研究人员测量了每个细胞与最近血管的距离,并发现血液蛋白与与血管距离的接近程度呈正相关。经过空间聚类,组织根据其与血管接近程度被划分为四个聚类。富集分析显示,距离血管较远的聚类1显示“氧化磷酸化”术语的富集,而距离血管较近的聚类2和4显示与血液相关的术语的富集。研究人员观察到在整个组织中 GAPDH 蛋白质的一致强度。蛋白质组学图显示了两种主要模式:与体素:血管距离呈正相关的蛋白质和与体素:血管距离呈负相关的蛋白质。研究人员确定了一些与血管相关的蛋白质,如Alpha-2-Macroglobulin和Aspartate beta,并表达出特定的空间分布。他们还注意到许多与细胞外基质相关的蛋白质的存在,这些蛋白质在空间上呈相关性,并富集于与 ECM 架构相关的通路。研究人员进一步分析了胶原蛋白和整合素蛋白质的空间丰度,显示组织中不同亚家族之间的异质性。与 ECM 相关的蛋白质显示出比非 ECM 蛋白质更高的空间聚类。总体而言,这项空间分析揭示了蛋白质的分布、营养/氧梯度以及肿瘤组织内细胞外基质(ECM)在肿瘤发展中的作用。
他们的结论
这项研究揭示了肿瘤内蛋白质的空间丰度模式,并确定了作为肿瘤边界的空间标记蛋白质。此外,该研究还揭示了免疫应答驱动的细胞外肿瘤基质蛋白质网络,并展示了深度地形蛋白质组学在重新定义组织生物学和病理学分子水平理解方面的潜力。该研究还讨论了当前空间分辨蛋白质技术的限制和挑战,并提出了未来研究的方向。