基于快速纳米孔测序AI术中诊断中枢神经系统肿瘤分型
J. de Ridder和同事们进行了一项研究,关于开发和使用一种名为Sturgeon的神经网络分类器,用于在手术中对中枢神经系统(CNS)肿瘤进行分类。他们最近在Nature上发表了他们的研究成果 (DOI:10.1038/s41586-023-06615-2)。该团队对目前确定精确肿瘤类型的方法存在的局限性进行了探讨,并考察了在手术过程中使用快速纳米孔测序获取甲基化分析可能带来的潜在好处。Sturgeon分类器被优化以处理稀疏数据,并在模拟的纳米孔测序运行中进行训练。研究表明,仅基于20-40分钟的测序时间,Sturgeon就能准确地对CNS肿瘤样本进行分类。在现实的手术操作环境中,Sturgeon通过在90分钟以内提供诊断结果,展示了其对手术决策的影响力。强调了精确可靠的诊断对于指导神经外科策略的重要性。纳米孔测序的使用带来了如低设备成本、小型设备以及即时数据获取的优势。尽管Sturgeon方法需要一定量的组织,但是成功的测试显示,它可能有助于防止神经并发症和需要进行额外手术的需求。
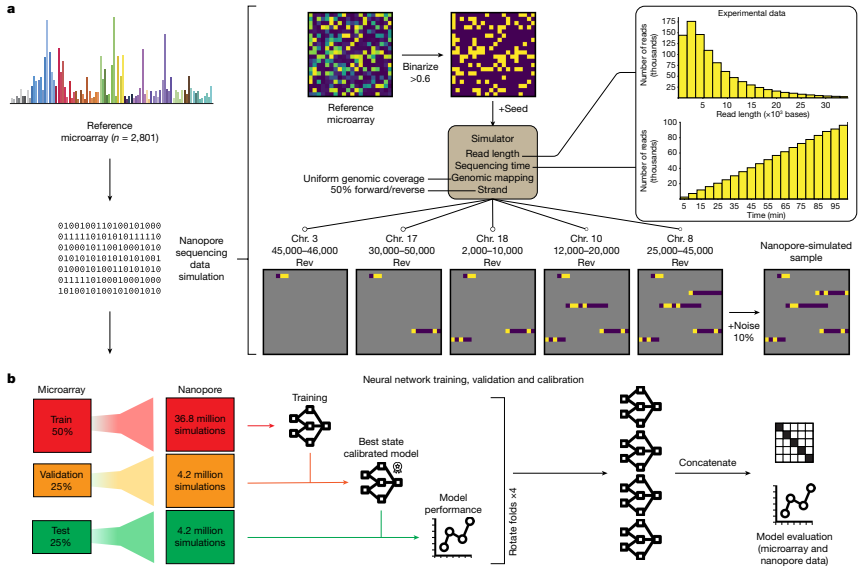
研究人员使用模拟的纳米孔测序数据来训练和验证他们的CNS肿瘤分类的Sturgeon模型。具体来说,他们使用了四个各自独立训练、验证和校准的神经网络。训练数据由Capper等人的参考数据集生成的模拟数据构成,该数据集被分成四份,同时保持原始类别的分布。研究人员使用两份数据来训练子模型,用一份数据来确定子模型表现最好的状态并进行得分校准,用最后一份数据来评估子模型的性能。神经网络首先使用0.6%到14%稀疏度的模拟进行预训练,然后使用0.6%到6.3%稀疏度的模拟进行最终分类器的微调。为了验证模型的性能,研究人员在留出的测试折叠上评估了其最终性能,每个样本为测试集贡献了6,000个模拟样本。他们评估了所有稀疏程度下的分类准确率和其他指标来评价模型的性能。研究人员还在日常诊断过程中获得的儿科甲基化分析上验证了他们的模型。此外,他们对公开可用的纳米孔测序数据进行了分类,这些数据来自GSE209865。总的来说,Sturgeon模型是通过使用模拟的纳米孔测序数据进行训练和验证的,包括来自Capper等人参考数据集的模拟以及公开可用的纳米孔测序数据。通过使用各种指标评估模型的性能,并在真实的儿科甲基化分析上进行验证。
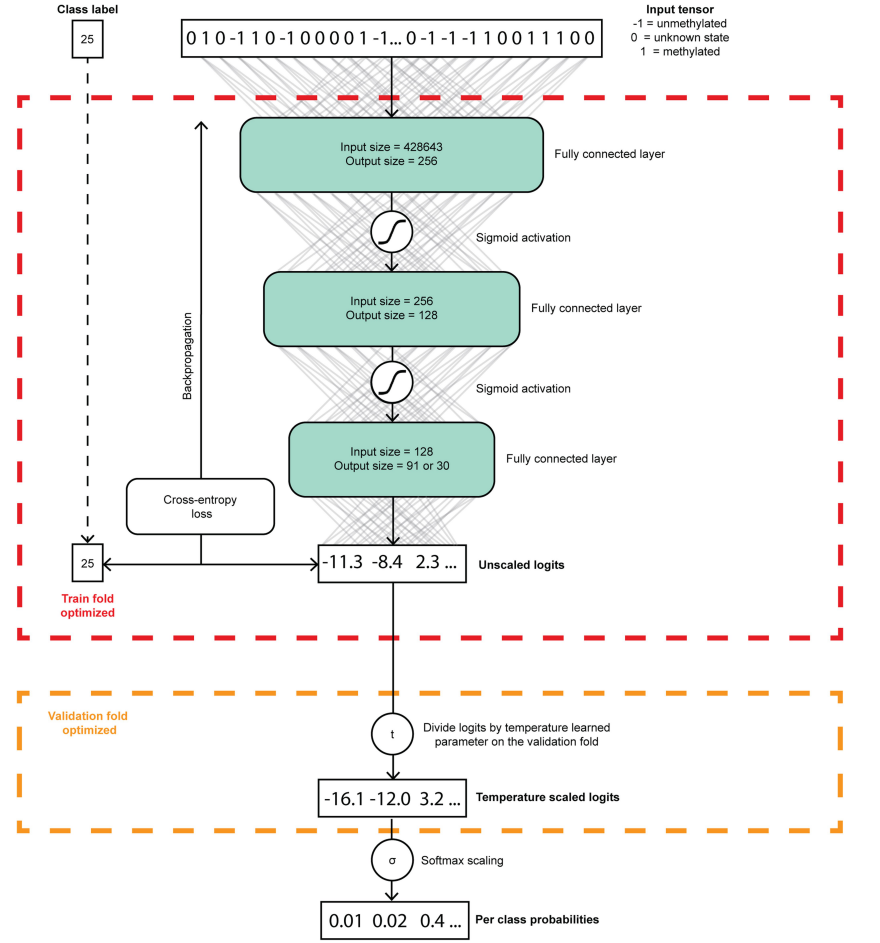
Sturgeon的训练数据集包括不同年龄(平均年龄为29,其中36%低于13岁)的病人。我们首先旨在进一步验证Sturgeon在儿科环境中的性能。为此,我们获取了94个基因组范围内的甲基化分析,这些分析是使用Illumina Infinium Methyla-tionEPIC v1.0(以下简称EPIC)阵列从在Princess Máxima Center (PMC) 进行CNS肿瘤切除手术的患者那里获得的。对于每个样本,公开可用的Heidelberg分类器 (v11b4) 被应用作为常规临床护理的一部分。这个分类器可以被认为是Capper等人分类器的更新版本。临床诊断的推荐临界值是0.84,该分类器对大多数样本(n = 68)达到了这个临界值。那些分类低于0.84的样本被认为根据其甲基化分析很难进行诊断,这可能是因为他们遇到了不常见的肿瘤类型,这些类型与之前注释的类别没有对应关系,或者是在遗传肿瘤预设疾病的背景下发生的肿瘤,或者是异质样本,或者是肿瘤纯度低的样本。对于每一个甲基化分析,我们模拟了500次纳米孔测序实验,考虑7种测序深度,总共进行了332,500次模拟的纳米孔测序实验,然后我们应用了Sturgeon分类器。
神经外科医生可以在手术中使用Sturgeon神经网络分类器来预测肿瘤的分类。Sturgeon分类器基于快速的纳米孔测序,这使得在手术过程中可以获得肿瘤的甲基化分析。这种方法克服了目前术前影像和术中组织学分析的局限性,这些方法可能并不总是能够提供确定或精确的肿瘤类型诊断。
Sturgeon分类器被优化以处理稀疏数据,并以模拟的纳米孔测序运行进行了训练。它可以在只有20-40分钟的测序时间基础上准确地分类CNS肿瘤样本。在现实的手术操作环境中,Sturgeon已经显示出能够在90分钟内提供诊断结果,从而影响手术决策。通过快速获得分子亚型诊断,神经外科医生可以做出更加明智的决策,关于肿瘤切除的程度,从而降低神经损伤和并发症的风险。
在Sturgeon方法中使用纳米孔测序提供了如低设备成本、小型设备以及即时数据获取的优点。虽然目前Sturgeon方法需要一定量的组织,但它已经成功地通过较小样本的测试。总的来说,在手术中使用Sturgeon分类器可以协助神经外科医生进行精确和可靠的肿瘤分类,指导他们的手术策略,可能防止神经并发症和需要进行额外手术的需求。